医院动态
CGGA数据库发布2000例脑胶质瘤组学数据 中国数据库面向全球研究者免费开放
近日,中国脑胶质瘤基因组图谱计划(CGGA, Chinese Glioma Genome Atlas; http://cgga.org.cn)数据库发布2000例中国脑胶质瘤样本的功能基因组学数据。经过15年的临床标本和组学数据积累,目前该数据库日臻完善,全部基因组数据向全世界研究者免费开放。据不完全统计,已有美国、欧洲多家知名研究机构近200篇SCI论文引用该数据库。
CGGA发起人和创建者,北京市神经外科研究所副所长、首都医科大学附属北京天坛医院神经外科副主任江涛介绍,此次公布的2000例中国脑胶质瘤样本的功能基因组学数据,涉及不同组织病理分类、不同WHO恶性度分级、原发/复发配对样本,功能组学数据涵盖全外显测序 (286例)、mRNA芯片(301例)及其测序(1018例)、microRNA芯片(198例)、以及DNA甲基化芯片(159例)。
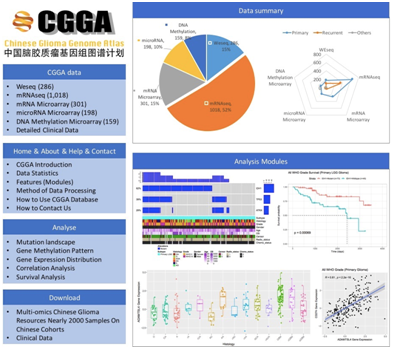
据介绍,这个数据库中包含了详尽的临床数据,包括患者性别、年龄、放疗和化疗情况、完整随访数据等,针对不同组学数据特点,研究团队还开发了不同的在线分析工具,包括脑胶质瘤突变图谱绘制、基因表达及其DNA甲基化的分布模式展示、相关性分析以及生存分析结果可视化等。
该数据库的建立有助于描绘中国人群脑胶质瘤的基因组及分子遗传学特征,探寻脑胶质瘤发生发展过程中的重要分子机制,为脑胶质瘤的分子分型和药物靶点研发提供指导,并为脑胶质瘤精准医学全链条的发展奠定基础。
“希望通过基因组学技术分析,全面绘制中国人群的脑胶质瘤基因组图谱,让国内外更多从事脑胶质瘤的研究者可以对数据库进行挖掘利用。”江涛说。
脑胶质瘤是成人最常见的颅内原发恶性肿瘤。2004年起,江涛开始致力于中国脑胶质瘤生物样本库的建立及临床随访数据的收集。在我国神经外科创始人王忠诚院士的指导下,2012年江涛团队启动CGGA。
依托于国家神经系统疾病临床医学研究中心、北京市神经外科研究所,以及首都医科大学附属北京天坛医院,目前CGGA数据库已经成为亚洲乃至全世界最大规模的脑胶质瘤医学信息工程。江涛表示,不断建设和完善的CGGA数据库,将对促进我国脑胶质瘤领域基础和临床研究水平的发展并提高我国癌症研究的国际影响力,最终推动脑胶质瘤新型诊疗模式的发展,让更多脑胶质瘤患者获益起到积极作用。
CGGA数据库代表性文章产出:
1.Hu H, Mu Q, Bao Z, Chen Y, Liu Y, Chen J, Wang K, Wang Z, Nam Y, Jiang B, Sa JK, Cho HJ, Her NG, Zhang C, Zhao Z, Zhang Y, Zeng F, Wu F, Kang X, Liu Y, QianZ, Wang Z, Huang R, Wang Q, Zhang W, Qiu X, Li W, Nam DH, Fan X, Wang J, Jiang T.Mutational Landscape of Secondary Glioblastoma Guides MET-Targeted Trial in BrainTumor. Cell. 2018 Nov 29;175(6):1665-1678.e18.
2.Bao ZS, Chen HM, Yang MY, Zhang CB, Yu K, Ye WL, Hu BQ, Yan W, Zhang W, Akers J, Ramakrishnan V, Li J, Carter B, Liu YW, Hu HM, Wang Z, Li MY, Yao K, Qiu XG, Kang CS, You YP, Fan XL, Song WS, Li RQ, Su XD, Chen CC, Jiang T. RNA-seq of 272 gliomas revealed a novel, recurrent PTPRZ1-MET fusion transcript in secondary glioblastomas. Genome Res. 2014 Nov;24(11):1765-73.
3.Zhang Y, Li J, Yi K, Feng J, Cong Z, Wang Z, Wei Y, Wu F, Cheng W, Samo AA, Salomoni P, Yang Q, Huang Y, Kang C, Jiang T, Fan X. Elevated signature of a gene module coexpressed with CDC20 marks genomic instability in glioma. Proc Natl Acad Sci U S A. 2019 Apr 2;116(14):6975-6984.
4.Shan X, Fan X, Liu X, Zhao Z, Wang Y, Jiang T. Clinical characteristics associated with postoperative seizure control in adult low-grade gliomas: a systematic review and meta-analysis. Neuro Oncol. 2018 Feb 19;20(3):324-331.
5.Wang Z, Hao Y, Zhang C, Wang Z, Liu X, Li G, Sun L, Liang J, Luo J, Zhou D, Chen R, Jiang T. The Landscape of Viral Expression Reveals Clinically Relevant Viruses with Potential Capability of Promoting Malignancy in Lower-Grade Glioma. Clin Cancer Res. 2017 May 1;23(9):2177-2185.
6.Yang P, Cai J, Yan W, Zhang W, Wang Y, Chen B, Li G, Li S, Wu C, Yao K, Li W, Peng X, You Y, Chen L, Jiang C, Qiu X, Jiang T; CGGA project. Classification based on mutations of TERT promoter and IDH characterizes subtypes in grade II/III gliomas. Neuro Oncol. 2016 Aug;18(8):1099-108.
7.Zhang C, Cheng W, Ren X, Wang Z, Liu X, Li G, Han S, Jiang T, Wu A. Tumor Purity as an Underlying Key Factor in Glioma. Clin Cancer Res. 2017 Oct 15;23(20):6279-6291.
8.Sun Y, Zhang W, Chen D, Lv Y, Zheng J, Lilljebjörn H, Ran L, Bao Z, Soneson C, Sjögren HO, Salford LG, Ji J, French PJ, Fioretos T, Jiang T, Fan X. A glioma classification scheme based on coexpression modules of EGFR and PDGFRA. Proc Natl Acad Sci U S A. 2014 Mar 4;111(9):3538-43.
9.Yan W, Zhang W, You G, Zhang J, Han L, Bao Z, Wang Y, Liu Y, Jiang C, Kang C, You Y, Jiang T. Molecular classification of gliomas based on whole genome gene expression: a systematic report of 225 samples from the Chinese Glioma Cooperative Group. Neuro Oncol. 2012 Dec;14(12):1432-40.
10.Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC. miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression. Neuro Oncol. 2012 Jun;14(6):712-9.













































 京公网安备111010602104056
京公网安备111010602104056